Homosügootne geenideletsioon
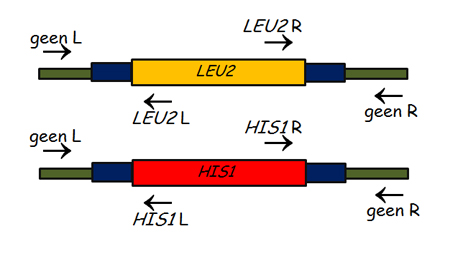
Candida albicans on diploidne organism. Seepärast nõuab deletsioonimutandi konstrueerimine kahe alleeli järjestikulist deleteerimist. Geeni deleteerimiskes kasutatakse lineaarseid DNA molekule, mis sisaldavad selektsioonimarkerit ning geenist ülesvoolu ja allavoolu jäävaid homoloogilisi järjestusi (joonis 7). Selektsioonimarkeritena kasutatakse auksotroofseid markergeene (näiteks HIS1, LEU2, ARG4, URA3) või antibiootikumide resistentsusgeene (näiteks SAT1).
Paremini töötavad auksotroofsed markergeenid, mis pärinevad teistest Candida liikidest (st. mitte-albicans markergeenid): C. dubliniensis HIS1, C. maltosa LEU2, C. dubliniensis ARG4. Nende markergeenide kasutamisel väheneb tunduvalt valede integratsioonide tõenäosus.
URA3 markergeeni kasutamine on problemaatiline, sest paljud C. albicans fenotüübid (näiteks morfogenees, virulentsus) sõltuvad URA3 ekspressioonitasemest.
DNA homoloogiliseks integratsiooniks C. albicans genoomi on vajalik vähemalt 60 nukleotiidi pikkuse, lookuse spetsiifilise järjestuse olemasolu. Tavaliselt kasutatakse palju pikemaid järjestusi, sest see tõstab integratsiooni efektiivsust. Deletsioonikasseti konstrueerimiseks kasutatakse kas PCR meetodit või traditsioonilist kloonimist.
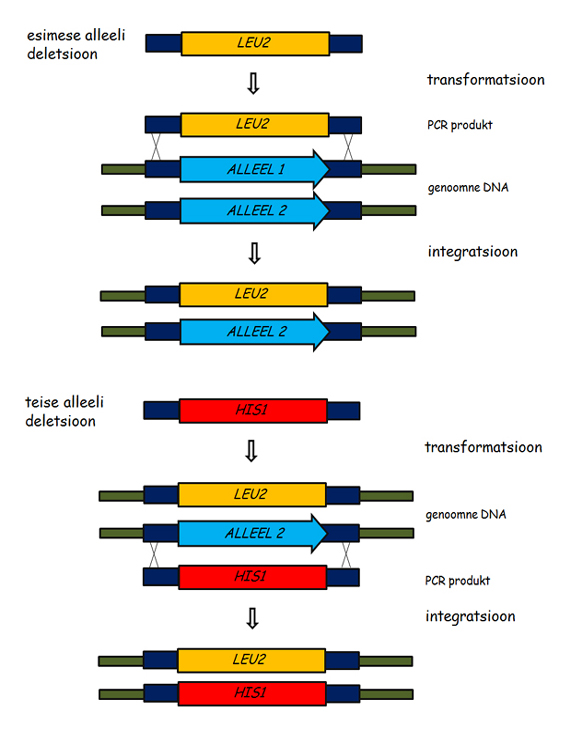
Transformantide selektsiooniks kasutatakse sobivat selektiivsöödet. Seejärel analüüsitakse PCR meetodit või Southern blot meetodit kasutades, kas deletsioonikasset on integreerunud õigesse lookusesse (joonis 8). Esimese alleeli deletsioonile järgneb teise alleeli deletsioon (joonis 7).